Diabetes Melito Tipo 1 (DM1): Uma predisposição genética
7 min de leitura
A diabetes melito tipo 1 (DM1) é a forma mais agressiva e é considerada o resultado de um processo autoimune específico contra as células-beta pancreáticas, ou seja, a formação de anticorpos que atacam as células beta, levando a deficiência de insulina.
O sistema imunológico é treinado para não atacar as células próprias. Eventualmente, essa “tolerância” imunológica falha e o resultado é uma doença autoimune, onde o sistema imunológico passa a atacar e danificar o tecido saudável.
Entendendo o HLA
O complexo principal de histocompatibilidade (MHC – Major Histocompatibility Complex) é um grande complexo gênico com múltiplos loci cujas moléculas apresentam antígenos proteicos às células do sistema imune, participando assim do processo de rejeição de tecidos estranhos ou próprios. Isso geralmente acontece em coordenação com o sistema imunológico que desencadeia uma resposta imediata contra esses corpos estranhos. A saber, nos humanos, esse conjunto gênico denomina-se HLA (Human Leukocyte Antigens).
A principal característica do complexo HLA é o seu polimorfismo, ou seja, múltiplas formas estáveis de um gene em uma população. Assim, diferentes indivíduos dentro de uma espécie apresentam formas levemente diferentes de cada gene HLA denominados alelos.
Numbers of HLA Alleles | ||||||
| HLA Class I Alleles | 17.191 | |||||
| HLA Class II Alleles | 6.716 | |||||
É importante saber que o HLA é herdado, ou seja, temos uma parte da mãe a outra do pai. O sistema HLA se localiza no braço curto do cromossomo seis humano. Em suma, os loci gênicos do HLA são agrupados em três classes; I, II e III, de acordo com sua estrutura, função e localização. A região de classe I é composta pelos loci HLA-A, B, C, que codificam as moléculas presentes praticamente em todas as células nucleadas. Já a região de classe II engloba os loci HLA-DR, -DP e –DQ, que codificam moléculas presentes, principalmente, na superfície de células imunocompetentes, incluindo macrófagos, células dendríticas, monócitos, linfócitos T ativados e linfócitos B. Enfim, a região de classe III possui genes que codificam componentes do complemento, como C4A, C4B e o fator B, além de conter genes para as enzimas 21-hidroxilase (CYP21), proteína do choque térmico (Hsp.70) e fatores de necrose tumoral
HLA Class I | ||||||
| Gene | A | B | C | |||
| Alleles | 5.266 | 6.537 | 5.140 | |||
| Proteins | 3.552 | 4.494 | 3.359 | |||
| Nulls | 286 | 232 | 236 | |||
HLA Class II | ||||||||||
| Gene | DRB1 | DQA1 | DQB1 | DPA1 | DPB1 | |||||
| Alleles | 2.581 | 183 | 1.718 | 5 | 132 | |||||
| Proteins | 1.834 | 76 | 1.151 | 2 | 52 | |||||
| Nulls | 78 | 6 | 72 | 0 | 1 | |||||
O entendimento desse polimorfismo é substancial para a compreensão de como o HLA é relacionado com doenças autoimunes, com a evolução das espécies, e, também, para a seleção de doadores em transplantes de órgãos.
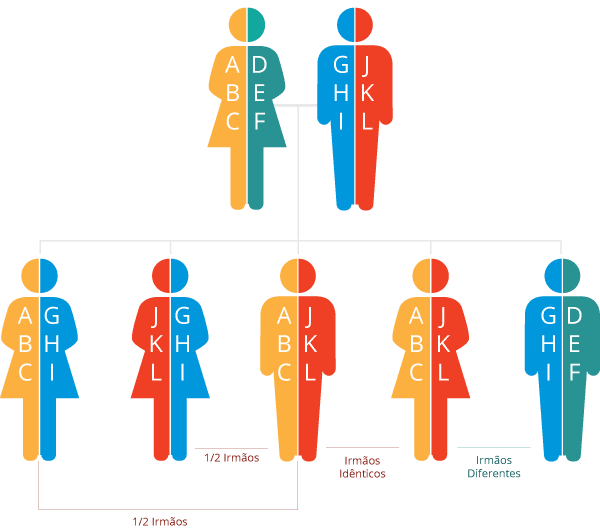
A Diabetes Melito e o HLA
Dois tipos de estudos têm sido utilizados para abordar a associação entre os marcadores de histocompatibilidade com as doenças como a Diabetes Melito (DM): estudos populacionais e os familiares. Nos estudos populacionais, as frequências dos antígenos ou dos alelos HLA, observadas em um grupo de pacientes não aparentados são comparadas com aquelas observadas em indivíduos de controle sadios. A ocorrência de associação é avaliada pela comparação das frequências dos marcadores de histocompatibilidade em pacientes e controle.
A Diabetes Melito (DM) tem sido considerada uma doença com herança poligênica complexa. Cerca de 20 genes podem estar associados com susceptibilidade à doença, mas apenas 13 apresentam evidências estatisticamente significantes de associação. A maior contribuição vem da região onde estão localizados os genes do HLA, situado no cromossomo 6p21, Já que contribui em cerca de 40% na susceptibilidade à doença (genes IDDM1).
Os loci DR e DQ são responsáveis por 40% a 50% do risco genético de desenvolver DM1. Como a região HLA exibe grande grau de desequilíbrio de ligação (ou seja, alelos DR e DQ não são associados randomicamente entre si), as associações do HLA com a doença são mais bem definidas pelos haplótipos do que pelos alelos. Assim, o maior risco é conferido pelos haplótipos de predisposição HLA-DQA1*05:01-DQB1*02:01 (chamado DQ2), geralmente herdado com DRB1*03:01 (DR3) e o haplótipo HLA-DQA1*-DQB1*03:02 (DQ8), herdado com DRB1*04:01 ou DRB1*04:05 (DR4).
Por outro lado, alguns haplótipos estão associados à proteção, particularmente HLA-DRB1*15:01/DQA1*01:02-DQB1*06:02 (DR2-DQ6), sendo o alelo DQB1*06:02 o principal responsável pela proteção do organismo à DM1.
No entanto, os fatores genéticos conhecidos, até o momento, podem ser responsáveis por no máximo 65% a 70% dos casos de DM1. A incidência da doença está aumentando, por isso outros fatores e sua relação com nosso organismo estão sendo estudados por especialistas. Principalmente, a influência hormonal, a influência de microrganismos presentes em nosso corpo e os fatores ambientais, como a exposição prolongada a produtos químicos, são alguns dos exemplos.
Métodos de detecção dos alelos HLA
Primordialmente o polimorfismo das especificidades HLA começou a ser descoberto através de métodos celulares. No entanto, os avanços na genética molecular, especialmente com a descoberta da reação em cadeia da polimerase (PCR – Polimerase Chain Reaction), possibilitaram a tipificação mais detalhada, com a identificação de diversos alelos. Portanto, os métodos moleculares mais utilizados para essa finalidade são o SSP (Sequence-Specific primers), SSOP (Sequence-Specific Oligonucleotides Probes) e SBT (Sequence-Based Typing).
O futuro da genética molecular: Sequenciamento de Nova Geração (NGS)
O Projeto Genoma, através do sequenciamento do DNA ou mapeamento do DNA, abriu diversas fronteiras e, acima de tudo, forneceu meios para identificar mutações e doenças genéticas específicas mais detalhadamente. Nesse sentido, permite determinar quais trechos de DNA contêm genes e quais trechos carregam instruções de regulação, ligando ou desligando os genes. Também abriu caminho para uma medicina mais personalizada, pois permite aos cientistas examinar até que ponto a resposta de um paciente a um medicamento é determinada pelo seu perfil genético.
Como resultado, essas conquistas exigiram o desenvolvimento e aprimoramento de novas tecnologias moleculares. A mais recente é o sequenciamento de nova geração, ou sequenciamento de segunda geração ou ainda Next-Generation Sequencing (NGS).
Através do NGS é possível realizar o sequenciamento de um genoma inteiro em questão de dias. A sua velocidade, capacidade de gerar dados em larga escala e precisão, a um custo inferior aos métodos tradicionais ganha, a cada dia, mais espaço. O princípio básico das plataformas de NGS está em sequenciar várias moléculas diferentes (ou vários fragmentos diferentes provenientes do mesmo genoma) em paralelo. Em cada reação é obtida uma única sequência de DNA, oriunda de um trecho específico. Isso é possível graças à integração de ferramentas de nanotecnologia, robótica e informática em aparelhos de alto desempenho.
Produtos
Referências:
- https://www.endocrino.org.br/o-que-e-diabetes/
- http://www.scielo.br/scielo.php?script=sci_arttext&pid=S1519-38292007000400002
- http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0004-27302003000500015
- http://www.scielo.br/pdf/abem/v52n2/04.pdf
- http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0004-27302008000200001
- http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0004-27302006000300005
- https://www.futurelearn.com/courses/diabetes-genomic-medicine/0/steps/10070
- https://www.biometrix.com.br/histocompatibilidade-o-que-e/
Lançamentos

Obrigado!
Aguarde os próximos passos, logo nossa equipe entrará em contato.
por gentileza, preencha os campos abaixo.