Fique por dentro de tudo!
Participe do nosso canal direto no WhatsApp para receber em primeira mão as novidades, lançamentos e oportunidades exclusivas da Kasvi.
7 min de leitura
Em reações PCR faz-se necessário trabalhar com DNA de boa qualidade. E conhecer a eficiência de cada processo na análise de DNA, tais como a extração, quantificação e purificação do DNA. É necessário extrair o DNA intacto para não quebrar as ligações, já que são estas as responsáveis pela informação genética. Qualquer que seja a técnica a utilizada, a qualidade nos processos envolvidos é fundamental.
Diversos tipos de amostras podem ser utilizadas para a extração, desde sangue até um único fio de cabelo. Entretanto, a amostra também irá determinar a qualidade do material extraído.
A quantidade de DNA purificado depende do tipo de amostra e do número de células presentes que pode variar, por exemplo, conforme a idade do paciente e seu estado de saúde e também devido as condições de transporte, armazenamento e idade das amostras. O volume utilizado é um fator muito importante.
Amostras frescas e congeladas de sangue terão um rendimento diferente. Amostras de sangue estocadas a 4°C por alguns dias ou congelado por semanas, podem ainda permitir isolamento de DNA. Porém, o rendimento e a qualidade do DNA podem diminuir devido ao armazenamento prolongado nestas condições. Certamente a qualidade e o rendimento de amostras se sangue fresco serão bem maiores do que amostras de sangue congeladas por anos.
Existem diversos kits para extração de DNA e RNA disponíveis no mercado, mas é imprescindível escolher qual é o kit ideal para atender ao que você necessita. É preciso levar em consideração a otimização do rendimento e a degradação do DNA ou RNA durante a extração, a sua eficiência em termos de custo, tempo e simplicidade da metodologia. Além disso, há a preocupação com componentes de baixo risco para o usuário, gerando ainda o mínimo possível de resíduos perigosos.
Alguns procedimentos básicos são realizados para isolar e purificar o DNA. São eles:
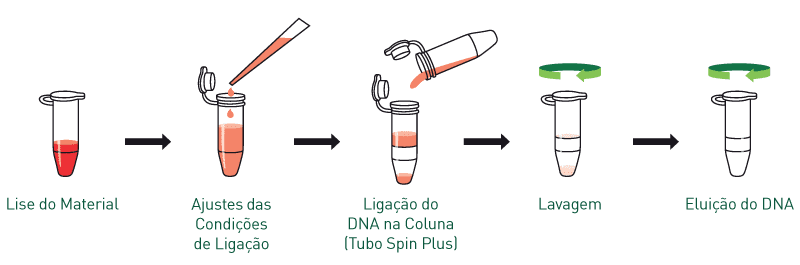
A extração de DNA e/ou RNA é o primeiro passo para a execução de diferentes procedimentos na Biologia Molecular. A escolha dos melhores produtos para essa etapa é fundamental para a qualidade final do diagnóstico.
Tampão de Lise T1 é a novidade da Kasvi na linha de reagentes. É a ferramenta ideal para uma extração e purificação manual simples, rápida e eficiente de diversos tipos de amostra. Proporciona até 13 protocolos de extração adicionais.
A quantificação envolve a estimativa da concentração do DNA obtida, que depende do tipo e quantidade de amostra disponível. Dentre os métodos disponíveis para quantificação tem-se a eletroforese em gel de agarose, que permite a resolução de ácidos nucléicos, um método comparativo, a utilização do fluorímetro, um equipamento que trabalha com alterações nas características de fluorescência na presença de DNA, um método quantitativo; e a espectrofotometria, realizada por medição da quantidade de luz absorvida pelo DNA.
A análise das bandas do gel, que correspondem a moléculas de DNA, é realizada aplicando-se também no gel dois tipos de soluções padrão, dependendo do objetivo da análise, que pode ser:
Nas extrações podem-se obter grandes moléculas, que incluem cromossomos inteiros ou cromossomos fragmentados. Amostras de boa qualidade formam bandas íntegras, enquanto amostras degradadas ou com presença de moléculas de RNA apresentam rastros ao longo do gel. A quantificação é feita pela comparação entre a intensidade da fluorescência das amostras extraídas com uma solução de DNA de concentração conhecida.
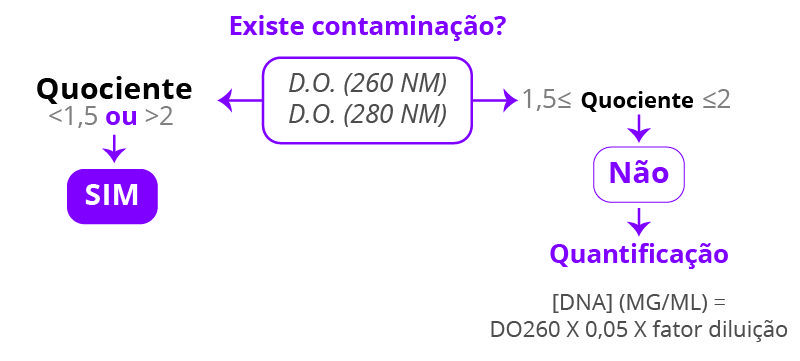
É capaz de determinar as concentrações médias dos ácidos nucléicos DNA ou RNA presentes em uma amostra, bem como sua pureza. A quantificação de DNA por espectrofotometria é realizada por medição da quantidade de luz absorvida pelo DNA em solução no comprimento de onda de 260 nm. Quanto maior for a absorção de luz nesse comprimento de onda, maior a concentração de DNA na solução. Assim, sabendo que o valor de absorbância (A) de 1,0 corresponde a uma concentração de 50 µg de DNA (fita dupla) por mililitro (mL), é possível calcular a concentração do DNA obtido das amostras.
É comum que as amostras de ácido nucleico sejam contaminadas com outras moléculas (proteínas, compostos orgânicos, outros). O benefício secundário da utilização de análise por espectrofotometria é a capacidade de determinar a pureza da amostra usando o cálculo de 260 nm: 280 nm. Valores entre 1,4 e 2,0 indicam pureza adequada das extrações de DNA, enquanto valores abaixo de 1,4 podem significar a presença de proteínas ou outros contaminantes.
Também conhecida como espectrofluorimetria é outro tipo de espectroscopia eletromagnética que analisa a luz emitida pelas moléculas fluorogênicas. Um método alternativo para avaliar a concentração de DNA e RNA é marcar a amostra com um marcador fluorescente, que é um corante fluorescente usado para medir a intensidade dos corantes que se ligam a ácidos nucléicos e fluorescem seletivamente quando ligados (por exemplo, brometo de etídio). Este método é útil para casos em que a concentração é muito baixa para avaliar com precisão pela espectrofotometria e nos casos em que os contaminantes que absorvem a 260 nm impossibilitam a quantificação exata por esse método. O benefício da quantificação de fluorescência de DNA e RNA é a melhor sensibilidade sobre a análise espectrofotométrica. Embora, esse aumento na sensibilidade acarrete o custo de um preço mais alto por amostra e um processo demorado de preparação da amostra.
